FANTOM изучает молекулярные процессы в клетках
 2796
2796 0
0
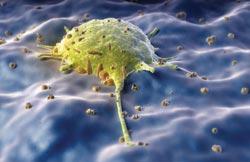
Целый каскад активирующихся в определенном порядке транскрипционных факторов ответственен за нормальное развитие лейкоцита.
Исследователи из консорциума PHANTOM использовали техники расшифровки нуклеотидных последовательностей матричных РНК, которые образуются в лейкемических клетках человека в ответ на присутствие во внешней среде форбол-миристил-ацетата. Данный реактив останавливает деление клеток, после чего они начинают дифференцироваться во взрослые белые элементы крови. Оценка продукции РНК проводилась в шести временных точках до начала дифференцировки и во время дифференцировки.
Первый анализ этих данных, опубликованный в журнале Nature Genetics [1], позволил реконструировать сеть молекулярных взаимодействий, которые приводят к активации и подавлению различных генов в процессе достижения клеткой конечной стадии специализации. Несмотря на то, что похожие модели были разработаны для дрожжей, проект PHANTOM на сегодняшний день – наиболее глубокое исследование регуляторных белковых сетей в клетках человека, как говорит Эран Сегал (Eran Segal), работающий над компьютерным моделированием биологических процессов в Weizmann Institute of Science в Израиле.
Участники консорциума считают, что разработанные ими методы сбора и анализа данных смогут помочь в моделировании реакций клеток на лекарственные препараты, а также направленной дифференцировки стволовых клеток в специализированные клеточные типы.
Консорциум PHANTOM включает более 100 независимых лабораторий, которые проводят параллельные эксперименты по анализу РНК в клетках, чтобы определить, какие именно гены активны на данной стадии дифференцировки. После оценки продукции РНК исследователи расшифровывали строение регуляторных участков активных генов, чтобы выяснить, какие транскрипционные факторы (белки, регулирующие экспрессию генов) работают в данный момент в клетке. После этого активность предполагаемых транскрипционных факторов была проверена с помощью экспериментов по блокированию их работы. Таким образом были получены высоко достоверные данные о работе в дифференцирующихся лейкемических клетках 52-х транскрипционных факторов.
Полученные данные говорят о том, что за дифференцировку клеток ответственна сложная сеть транскрипционных факторов, при этом нет какого-то одного «самого главного» фактора, определяющего направление дифференцировки.
Чувствительность использованных методов также позволила исследователям выявить дополнительные процессы – например, продукцию коротких молекул РНК (не более 18 нуклеотидов в длину), которые транскрибируются с участков ДНК, расположенных между генами [2]. Пьеро Карнинчи (Piero Carninci) из Японского Института Физики и Химии (RIKEN) в Йокогаме использовал базу данных PHANTOM, а также некоторые другие, для анализа молекул РНК, образующихся на базе ретротранспозонов – некодирующих участков ДНК, функции которых пока неизвестны. Ретротранспозоны способны свободно перемещаться в пределах генома благодаря специальному молекулярно-генетическому механизму. Интересно, что характер их активации тесно связан со стадией развития клетки и специфичен для разных клеточных типов [3]. Карнинчи считает, что подобные исследования помогут разобраться, какую функцию выполняют ретротранспозоны в клетке.
Подобные исследования, несмотря на то, что требуют огромных денежных и временных затрат, должны, как считают ученые, обязательно окупиться. Тем не менее, получение и анализ этих данных – только первый шаг очень долгого пути.
Литература:
1. The FANTOM Consortium and the Riken Omics Science Center Nature Genet. advance online publication doi:10.1038/ng.375 (2009).
2. Taft, R. J. et al. Nature Genet. advance online publication doi:10.1038/ng.312 (2009).
3. Faulkner, G. J. et al. Nature Genet. advance online publication doi:10.1038/ng.368 (2009).
По материалам:
NatureNews